Una técnica 'casera' de PCR para identificar las mutaciones
Investigadores del ISPA y del HUCA emplean un método «más sencillo, barato y rápido» para definir las cepas del coronavirus en los positivos
El bioquímico estadounidense Kary Mullis describió hace ya 40 años la técnica que ahora ya forma parte de nuestro vocabulario diario: la de la PCR ... (acrónimo de 'reacción en cadena de la polimerasa'). Es la forma más 'popular' de diagnosticar SARS-CoV-2, claramente identificada por la imagen de esa toma de muestras usando una torunda en la nariz. Una técnica ampliamente empleada en biología, no solo para el diagnóstico virológico, sino también para detectar otras bacterias o parásitos.
Nuestra imagen se frena ahí, en la propia toma de la muestra. Pero la clave de esa PCR se produce en el laboratorio, donde se amplifica el genoma del virus para detectar y cuantificar la presencia del bicho. En general. Sin apellidos. Si ya queremos hablar de variantes, entran en juego otras técnicas y de ellas saben mucho en la sección de Virología del Servicio de Microbiología del HUCA, algunos de cuyos miembros pertenecen además al Grupo de Microbiología Traslacional del ISPA, que coordina José Antonio Boga. Él, junto con el jefe de la sección, Santiago Melón, y las facultativas Marta Elena Álvarez-Argüelles y Susana Rojo, participan en investigaciones sobre este coronavirus. «Desde su aparición en China, el virus ha cambiado y ha ido acumulando mutaciones en su genoma. De hecho, las cepas que circularon en Asturias en la primera ola ya eran diferentes de la original de Wuhan», explica Boga. Y de esa base de datos creada con las numerosas variantes, ¿cuáles preocupan? «Aquellas que pueden suponer un problema porque sean más contagiosas, y/o más patogénicas, o bien, importante, se escapen a las vacunas. Las actuales o las futuras».
Llegados a este punto hay que preguntarse: ¿la PCR original sirve para determinar la clase de coronavirus que tenemos? La respuesta es no. Lo detecta, pero no define la variante. Entonces, ¿qué hay que hacer? «La forma de caracterizar esas variantes es mediante secuenciación del genoma completo del virus con técnicas de nueva generación. Hay que tener en cuenta que estamos antes uno de los virus de ARN más grandes de la naturaleza: tiene 30.000 bases en fila». Existe, además, una indicación del Ministerio de Sanidad para desarrollar una «vigilancia a tiempo real» y, en Asturias, cada semana se secuencian en el HUCA, con la colaboración del Servicio de Genética, unas 30 muestras positivas –porcentaje muy pequeño respecto al total de positivos semanales–. Es una técnica muy efectiva, pero cara (puede llegar a costar hasta 150 euros la muestra), lenta, y laboriosa (los resultados tardan una semana, y «dando cierta ventaja a las variantes»).
Método propio
El grupo del ISPA pensó, ante estos 'inconvenientes', en la posibilidad de utilizar un método diferente, en paralelo a la propia secuenciación, que permitiera esa detección de variantes «de forma sencilla, rápida y relativamente más barata, permitiendo hacer un cribado de variantes en todos los positivos, y no en unos pocos». ¿Cómo lo consiguieron? Modificando una técnica ya existente, que es una variante de la propia PCR que se usa para la detección del SARS-CoV-2 para poder localizar ciertas mutaciones.
Intentamos explicarlo, con la ayuda de José Antonio Boga, de una manera sencilla (ver esquema). La PCR original para el diagnóstico tiene una única sonda que se 'pega' (positivo) o no se 'pega' al coronavirus. Así, ya tendríamos un diagnóstico, pero no podríamos distinguir la variante que nos ha contagiado. Para saberlo, aplicaríamos, una segunda PCR denominada de 'discriminación alélica' que «distingue cambios en los nucleótidos, discriminando variantes».
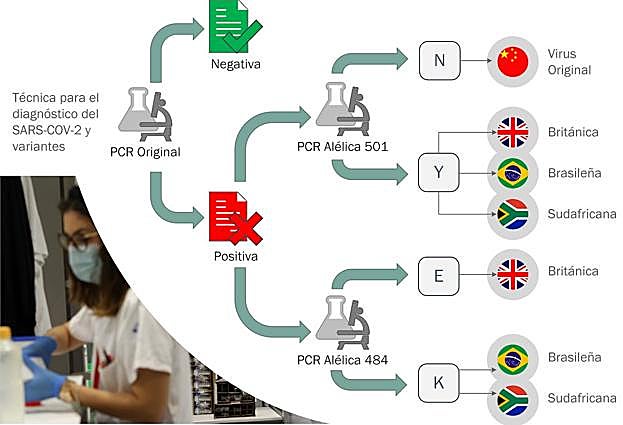
Recordamos que las tres variantes que más preocupan en estos momentos son la británica, que copa casi el 100% de los casos actuales en Asturias; la brasileña, con más de una docena de casos; y la sudafricana. Las tres se caracterizan porque tienen una mutación, la variante 501, con lo cual con la PCR alélica 501 podemos saber si tenemos una mutación del virus (Y). Pero no cuál de ellas es. El 'apellido' a nuestra variante se lo daremos con la otra PCR alélica, la 484, que distinguirá entre la cepa británica (E) o la brasileña/sudafricana (K). Actualmente, se está desarrollando otra PCR más, basada en la posición 417, que distinguirá entre las dos últimas.
José Antonio Boga explica las ventajas de ambas técnicas. Con la propia del PCR del ISPA, además de ser más barata, más rápida (la respuesta llega en 24 horas) y menos laboriosa, se pueden estudiar todas las muestras positivas. ¿Inconveniente? Que solo busca mutaciones puntuales y conocidas. La técnica de la secuenciación masiva, por su parte, permitiría distinguir si aparece una mutación nueva que no fuera ni la británica, ni la brasileña, ni la sudafricana, pero, como ya se ha dicho, es más cara, lenta y laboriosa. Cabe recordar que por las manos de estos profesionales pasan diariamente 1.500 muestras (no todas son positivas), con picos de hasta 5.000.
En este punto de la información es casi obligado preguntar al investigador:
–¿Se han detectado variantes que escapan a la inmunidad que supone la vacuna?
–Hacen falta más estudios. Algunos estudios de seroneutralización (donde se mide la capacidad del suero de una persona inmunizada con una determinada vacuna de neutralizar diferentes variantes del virus) parecen demostrar que esa capacidad es menor si enfrentamos esos sueros a las variantes de importancia, especialmente la sudafricana y brasileña. No hay que alarmarse, pero debemos de ser conscientes de la necesidad de una rápida vacunación a nivel mundial que corte la aparición de variantes nuevas. Además, se podrían desarrollar nuevas vacunas basadas en las actuales dirigidas contra estas variantes de escape. Quizá vayamos a un escenario tipo gripe en el que se necesitarían vacunaciones periódicas (quizá anuales) de vacunas diferentes a grupos de riesgo.
El investigador del ISPA realiza una reflexión final. Para empezar, no es la primera pandemia a la que se enfrenta el mundo. «Hace cien años fue la de la gripe, pero más cerca, en 2009, hubo la cepa gripal H1N1. El virus del Zika estuvo ahí, y el ébola es frecuente que nos dé un susto». A ello se une que estamos ante el séptimo coronavirus que afecta a los humanos: cuatro son leves y circulan habitualmente; el SARS-CoV, que hubo en China en 2002; y el MERS-CoV, de Oriente Medio. «Es un toque de atención que nos da la naturaleza. Hay que estar preparado y aprender, porque habrá que afrontar situaciones similares en un futuro más o menos cercano», señala Boga.
¿Tienes una suscripción? Inicia sesión